【解决方案】马尔文帕纳科为药物筛选按下快进键!
时间:2022-05-20 阅读:1056
Hot
马尔文帕纳科为药物筛选按下快进键!

利用生物物理片段筛选“甜蜜点”
发现抑制BRPF1 的新化学型
马尔文帕纳科为药物筛选按下快进键!
基于化合物库的有效采样是片段筛选命中 (fragment screening hit) 识别范式的核心。通常片段分子越小,采样效率越高。对于小分子和靶蛋白之间弱结合力的检测能力决定了片段分子可以设计的大小。
CLS*的片段筛选平台
马尔文帕纳科旗下 Concept Life Sciences 和 Creoptix 联手开发了一个*的片段筛选平台。该平台将专门构建的片段分子库与光栅耦合干涉 (GCI) 技术相结合,通过在生物物理“甜蜜点” (Sweet Spot) 进行筛选操作,可在几天内确定有效的片段命中 (fragment hits),从而为片段命中优化提供理想的起点,并为候选药物提名提供了更快的途径。
本文报告了使用该平台进行片段分子筛选,并识别了能有效结合 BRPF1 溴结构域的新片段。
BRPF1
Bromodomains 溴结构域是进化上保守的蛋白质-蛋白质相互作用模块,可识别组蛋白乙酰化赖氨酸。溴结构域和 PHD 指蛋白 1 (BRPF1) 是单核细胞白血病锌指 (MOZ) 组蛋白乙酰转移酶的亚基。它通过多个表观遗传阅读器结构域调节基因转录,包括*的双 PHD 和锌指组装、溴结构域和 C 末端 PWWP 结构域(如图 1所示)。
BRPF1 被认为是肝细胞癌[1]和急性髓细胞白血病[2]的治疗靶点,目前这种侵袭性癌症的成人 5 年生存率不到 30%[3]。
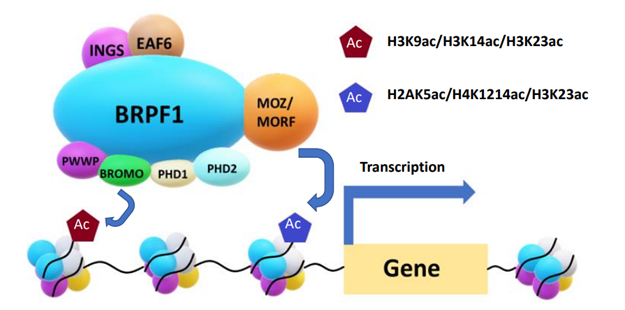
图 1. MOZ/MORF 组蛋白乙酰转移酶复合物介导 H3K9、H3K14 和 H3K23 乙酰化以激活基因。BRPF1 包含两个植物同源域 (PHD) 手指的乙酰阅读器结构域,它们由一个锌指节 (PZP 结构域)、一个溴结构域和一个脯氨酸-色氨酸-色氨酸-脯氨酸 (PWWP) 结构域隔开。
近年来报道了一系列 BRPF1 抑制剂,包括 GSK6853[4]和 NI-57 [5](图 2)。高通量片段对接 [6] 和基于配体的筛选 [7] 也被用于鉴定其他化学型 ,并在 X 射线结构的辅助下对其进行优化(图 2)。但是目前尚未有选择性的 BRPF1 抑制剂进入临床研究,所以能抑制这一靶点的新化学型仍是研究热点。

图 2. 左:选择性 BRPF1 抑制剂的化学结构。右图:BRPF1与片段分子结合的 X 射线结构 (pdb = 5EQ1)。BRPF1 以绿色带状表示,其表面由位于酰基结合域的残基侧链构成碳原子为绿色,氧原子为红色,氮原子为蓝色。片段分子以棒状表示。
生物物理“甜蜜点”
Sweet Spot
据新文献估计,化学宇宙已经由多达 11 个重原子、1420 万个化合物增长到了多达 17 个重原子、超过 1660 亿个化合物[8]。这种指数增长体现了片段分子大小的增加对化学领域的影响,而片段库的过分庞杂并不利于基于片段的药物设计 (FBDD) (图 3)。伴随着FBDD的发展,涌现出了多种生物物理技术能够检测到片段分子与靶点之间微弱的结合。因此,一个有效的筛选活动应能在筛选化合物时,可以最大限度的在生物物理技术可以检测到的极限范围内进行采样,即片段分子的生物物理“甜蜜点”(图 3)。

图 3. 化学宇宙的演变(橙色)、找到筛选命中的机会(灰色)和典型的亲和力(蓝色)与重原子数量的关系。虽然典型的商业片段库(紫色)倾向于集合较大的化合物,但通过高灵敏的生物物理技术,在生物物理“甜蜜点”(绿色)范围内,可以对更小的片段分子进行更有效的筛选。
生物物理片段库构建

图 4. 片段库构建流程:从 55,000 个 CLS 化合物集合到最终 1,133 个选定片段。该过程包括一个生物物理 MPO 对匹配生物物理“甜蜜点”的片段进行分类,以最大限度地提高多样性,并基于溶解度和纯度对化合物质量进行严格过滤和筛选。
最da化的化合物多样性

图 5.(A)CLS化合物库的主要成分分析。散点之间间隔越远,相应片段的结构越多样化(颜色对应于簇 #),(B)惯性矩阵图表示 CLS 片段分子的杆状、片状和球形形状(颜色对应于簇 #),(C ) 结构多样性由在不同 Tanimoto 指数下属于 1 到 5 个簇的化合物数量表示,(D) 1,133 个 CLS 片段的每个主要chemical handles的百分比。
片段命中类库
Fragment Hit-Like Library
在过去 20 年的 FBDD 研究中,已报告的片段命中 [9] 和商业片段集合的物理化学特征之间出现了明显的二分法,这通常由 3 规则 (Ro3) [10] (图 6) 支持 。
通过瞄准生物物理“甜蜜点”,CLS 生物物理片段集合(图 6)不仅可以有效地对可用的化合物库进行采样,而且还显示了与报告的片段命中非常匹配的物理化学特征。

图 6. Giordanetto 等人报道的 486 个片段命中的物理化学特征[9](绿色)、典型的商业片段集合(橙色,从 11 家供应商的 120,000 多个片段中收集)和Concept Life Sciences构建的生物物理片段集合(蓝色)。
生物物理筛选

图 7. SPR 和 GCI 筛选工作流程。GCI 技术使用Creoptix WAVE delta 系统(图片),该系统可实现简单且省时的片段筛选。
使用 waveRAPID®
进行动力学筛选

Creoptix WAVE Delta
GCI 分子间相互作用分析仪
这是一种测量动力学的新方法, 不依赖于梯度浓度滴定。waveRAPID®脉冲式注入单一浓度样品,通过增加样品在传感器表面的滞留时间,即可从单个样品孔中获得动力学(图 8)。由于 GCI 的高灵敏度,片段命中可以根据更丰富的数据进行排名,并根据动力学速率常数而不是单独的亲和力来选择命中。

图 8. 传统动力学检测与 waveRAPID® 比较。在典型的配体结合分析中(左),分析物以增加的浓度加入,每次注入的持续时间一致。在waveRAPID ®分析中(右),该系列由一个循环组成,其中只有一个被解离部分中断的结合阶段和一个最终解离阶段。模型拟合用解离阶段来完成。
实验设计和初步筛选数据
-
基于动力学速率常数拟合、Rmax 和 KD 的命中选择
-
使用 2 个传感器芯片、1100 个移液器吸头、4 个样品板和 2.1L 缓冲液,运行时间为 54 小时,可提供完整的动力学信息。


图 9:waveRAPID® 实验结果。(A) 在基于GCI技术的 WAVE delta 4 个不同通道上设置的实验设计。(B) 使用 WAVE control工作站 的 Direct Kinetics 引擎(黑色)将waveRAPID® 传感图(红色)拟合到一对一绑定模型的示例,并提供 Rmax、ka(结合速率)、kd(解离速率)和 KD( 解离平衡常数)。(C) 生成的 waveRAPID® 速率图,显示每个片段结合剂的 kd vs ka(颜色为 kd )。产生的 KD 范围用虚线突出显示。
参考文献:
(1) Cheng, C.LH., et. al. Bromodomain-containing protein BRPF1 is a therapeutic target for liver cancer. Commun. Biol. 4, 888 (2021)
(2) Shima, H., et. al. Bromodomain-PHD finger protein 1 is critical for leukemogenesis associated with MOZ-TIF2 fusion. Int J Hematol. 99, 21-31 (2014).
(3) cancerresearchuk.org/about-cancer/acute-myeloid-leukaemia-aml
(4) Bramborough, P., et. al. GSK6853, a Chemical Probe for Inhibition of the BRPF1 Bromodomain. ACS Med. Chem. Lett. 7, 6, 552–557 (2016)
(5) Igoe, N., et. al. Design of a Biased Potent Small Molecule Inhibitor of the Bromodomain and PHD Finger-Containing (BRPF) Proteins Suitable for Cellular and in Vivo Studies. J. Med. Chem. 60, 2, 668–680 (2017)
(6) Zhu, J., et. al. Twenty Crystal Structures of Bromodomain and PHD Finger Containing Protein 1 (BRPF1)/Ligand Complexes Reveal Conserved Binding Motifs and Rare Interactions. J. Med. Chem. 59, 11, 5555–5561 (2016)
(7) Zhu, J., et. al. Structure-based discovery of selective BRPF1 bromodomain inhibitors. Eur. J. Med. Chem. 155, 337-352 (2018)
(8) Ruddigkeit, L., et. al. Enumeration of 166 billion organic small molecules in the chemical universe database GDB-17. J. Chem. Inf. Model., 26, 52, 2864-75 (2012)
(9) Giordanetto, F., et. al. Fragment Hits: What do They Look Like and How do They Bind? J. Med. Chem. 62, 3381−3394 (2019)
(10) Congreve, M., et. al. A 'Rule of Three' for fragment-based lead discovery. Drug Discov. Today 8, 876–877 (2003
