Gibson Assembly原理
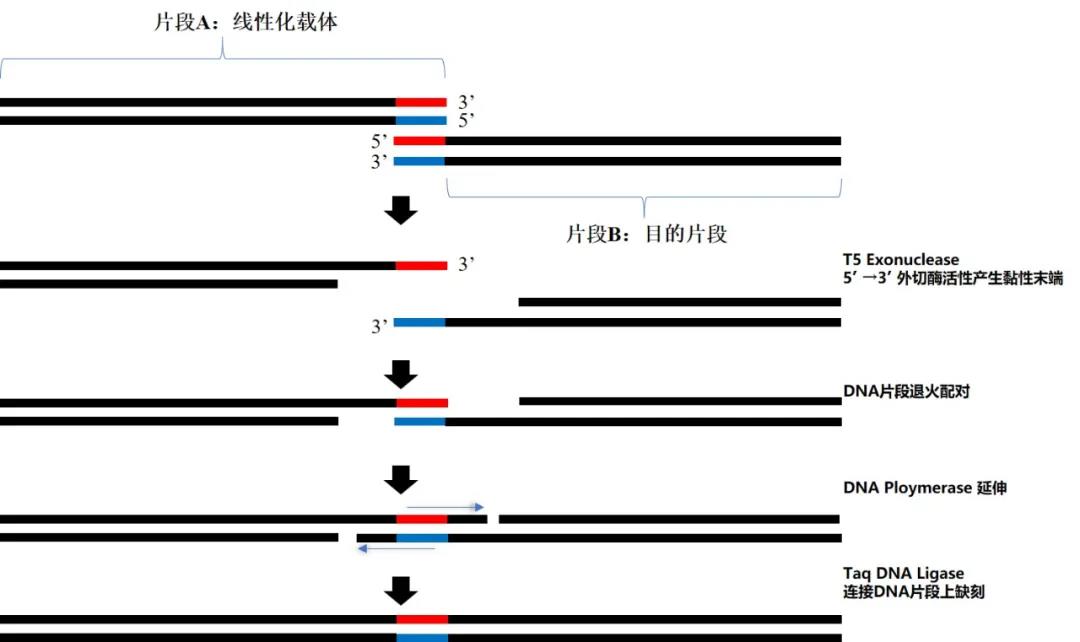
Gibson Assembly原理示意图
In-Fusion Cloning利用了In-Fusion酶能够识别3’末端碱基,具有3’→5’外切酶活性的特点。通过In-Fusion酶产生5’黏性末端,互补重叠片段退火配对;再将重组片段转化到感受态细胞内,重组片段序列中的缺刻在菌体内进行连接,获得重组序列。

无缝克隆的优势
不受酶切位点限制
适用于任意载体、任意片段
一步连接1或多个片段,连接多片段耗时短
插入目的片段两端不会添加额外序列(如保护碱基)

无缝克隆实验关键影响因素
片段质量
确保DNA片段无核酸酶及其它酶污染、无乙醇等残留;
纯度正常:A260/A280值在1.6-1.9、A260/230值在2左右、紫外吸收峰图正常;
浓度正常:插入片段与线性化载体浓度高于50 ng/μl,会使重组效率提高;
若模板浓度低、纯度低,则连接效率降低。
载体线性化方法
线性化载体可以选择反向PCR或酶切的方法:
可根据在载体插入位置是否有合适的酶切位点选择具体方法。因Gibson Assembly反应体系内无双链DNA连接酶,不会发生载体自连,线性化载体不需要进行末端去磷酸化处理。酶切制备线性化载体建议使用双酶切,且酶切后采用胶回收的方法回收产物,这样利于降低假阳性。
若没有合适酶切位点或者多次酶切制备线性化载体均不成功,可以选择反向PCR扩增,但不适合较大质粒,如超过8 kb。
引物设计
目的片段引物设计:目的片段引物是由目的片段两端序列加同源臂序列组成,同源臂长度设置在16-45 bp,Tm值在58–65°C。同源臂越长,有利于重组置换,但可能会影响目的片段扩增;
载体引物设计:确定目的片段插入位置,推荐借助Snapgene软件设计反向PCR引物。
PCR扩增
反向PCR制备线性化载体及扩增目的片段时需要使用高保真酶;
制备线性化载体后使用Dpn I内切酶消化环状质粒模板,降低假阳性克隆。
插入片段与线性化载体摩尔比
10 µl 反应体系,载体与插入片段总加入量建议在 0.01-0.3 pmol。插入片段与线性化载体的最佳摩尔比为 1:1-4:1;片段与片段的摩尔比为1:1。
推荐使用GenStar 克隆连接反应体系计算工具,快速计算载体与目的片段使用量。
感受态转化效率
插入片段与载体全长超过10 kb,属于长片段克隆,应使用高效感受态;如:转化效率大于108 cfu/μg DNA的感受态细胞。
感受态细胞与重组质粒用量比例不小于10:1。
只要注意以上几点,高效无缝克隆实验轻松get。
什么?不清楚无缝克隆实验具体怎样操作?
GenStar 无缝克隆产品列表




