从1985年至今的30多年时间里,PCR分析经历了三代技术的发展。diyi代传统PCR技术,采用琼脂糖凝胶电泳的方法对PCR产物进行定性分析。第二代荧光定量PCR技术,通过在PCR反应体系中加入荧光基团,利用荧光信号的积累实时监控PCR进程,后用Cq值对基因进行定量分析。第三代数字PCR技术,通过将PCR反应液进行有限稀释,实现核酸分子的单分子扩增,终采取终点法检测阳性微滴的荧光信号,有荧光信号的微滴判读为 1;没有荧光信号的微滴判读为 0,因此该技术被称为数字PCR。
PCR技术在不断地蜕变却从未淡出我们的视野,如今已经渗透到分子生物学领域的各种研究层面。尤其在今年大流行疾病中,展现了PCR技术的强大之处。那在科学研究中,我们该如何选择传统PCR 、荧光定量 PCR与数字 PCR呢?
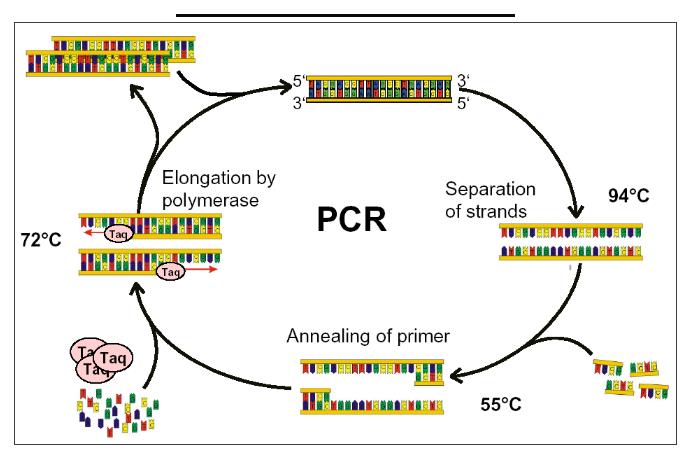
首先我们要清楚,三代PCR技术所有的基础源于PCR反应的基本原理和PCR反应的3个重要阶段,分别是指数期、线性期和平台期。
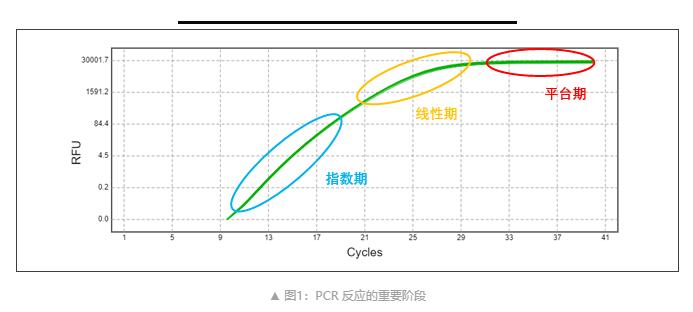
指数期
指数期内,每个循环PCR产物量大约增加1倍(假定 100%反应效率),该阶段的扩增反应具有高度特异性和jingque度。
线性期(高变异性)
随着PCR反应体系成分的消耗,其中的一个或者多种成分限制反应,导致反应开始减缓,并且每个循环的PCR 产物不再加倍。
平台期
反应停止,不再产生更多的产物。因为每个样品具有不同的反应动力学,所以每个反应将在不同的时间点进入平台期,平台期内可以看到这些差异。
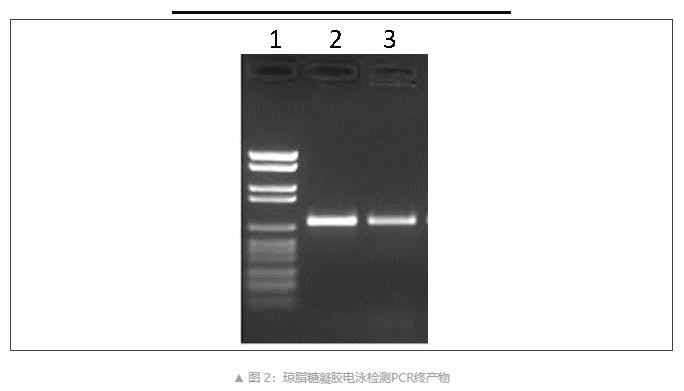
传统 PCR
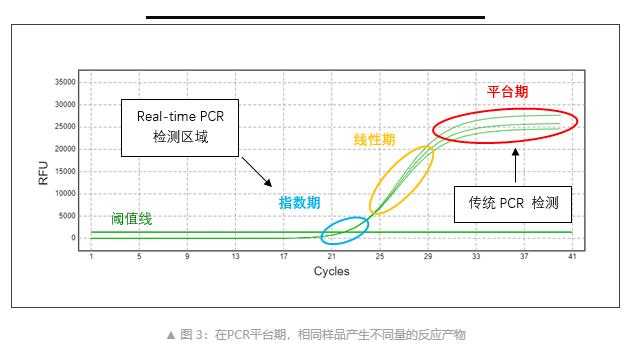
传统PCR通过琼脂糖凝胶电泳对PCR终产物进行分析(图2),它的局限性就在于只能做定性分析。在图3中,3个反应孔含有相同量的 DNA模板,但到达平台期后产生的荧光信号却不同,说明扩增产物的量存在不同(反应动力学变化所致)。这也表明了传统PCR平台期测量时结果存在变异,产出的DNA量不一定能反映初情况,所以无法进行定量。
荧光定量 PCR
同样在图3中,发现在指数期内,3个反应孔的扩增曲线*重合,说明指数期内进行检测将更加jingque,可实现更加准确的定量。阈值线是扩增产物荧光强度超过背景时的一个荧光强度值,样品达到此荧光强度值所经过的循环数称为Cq值。通过比较未知浓度的样品与一系列标准品的Cq值,可以jingque测定未知反应中的模板 DNA数量。
数字 PCR
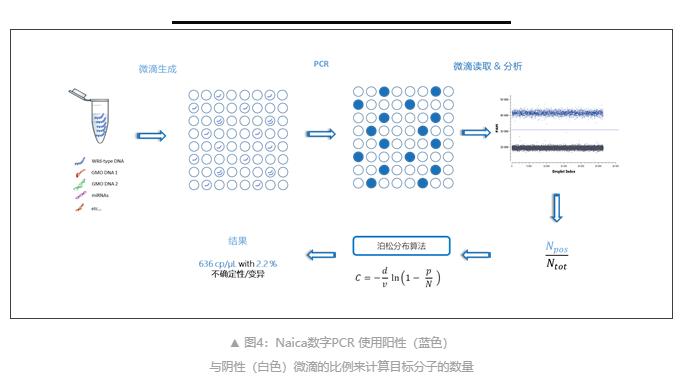
数字 PCR 是通过将样本DNA随机分布至独立的反应单元中,每个反应单元中包含或者不包含1个或多个拷贝的目标分子,所有独立的反应单元进行平行扩增后,对每个反应单元的阴性或者阳性荧光信号进行检测并进行统计学分析,就可以计算出原始样本的拷贝数,无需参考标准品或内源性对照品,也不需要标准曲线。
传统PCR 、荧光定量 PCR与数字 PCR的选择

深蓝云生物科技为您提供美国Azure Cielo 3/6通道荧光定量PCR系统和法国Stilla Naica全自动3色/6色微滴芯片数字PCR系统。
Azure Cielo™实时荧光定量PCR系统
来自于美国Azure Biosystems公司,为您提供Azure Cielo-3通道和Azure Cielo-6通道,可根据实验需求灵活配置。这款产品采用了高能LED作为光源系统,可保证光源强度高,光源一致性好;高品质的帕尔贴温度模块作为温控系统,升降温速率快,可设置12列跨度30°C的温度梯度;zhuoyue的CMOS拍照+光纤信号传输作为检测系统,CMOS检测灵敏度高,光纤传输速度快,无光损失和干扰。Azure Cielo™实时荧光定量PCR系统可为您的科学研究提供高jingzhun度、高灵敏度和高可靠性的实验结果。

Naica™微滴芯片式数字PCR系统
法国Stilla Technologies Naica数字PCR平台是实现高灵敏DNA/RNA定量的技术工具,只需一步移液操作,即可自动生成单层平铺的微滴,同一反应液进行多基因的单分子模板扩增,通过三色或六色荧光通道检测,自动QC质控,识别多色荧光中有效的阴阳性微滴,从而达到目的DNA/RNA的高jingzhun定量。同时提供原始数据、单微滴追溯等功能,确保数据真实可靠。

参考文献:
1. Sykes PJ, Neoh SH, Brisco MJ, Hughes E, Condon J, Morley AA. Quantitation of targets for PCR by use of limiting dilution. Biotechniques. 1992;13(3):444-449.
2. Vogelstein B , Kinzler K W . Digital PCR[J]. Proceedings of the National Academy of ences of the United States of America, 1999, 96(16):9236-9241.
3. Heyries KA, Tropini C, Vaninsberghe M, et al. Megapixel digital PCR. Nat Methods. 2011;8(8):649-651.
4. Dingle TC, Sedlak RH, Cook L, Jerome KR. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances. Clin Chem. 2013;59(11):1670-1672.
5. Whale AS, Huggett JF, Cowen S, et al. Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation. Nucleic Acids Res. 2012;40(11):e82.




