12597ESHieff NGS® In-Situ DNA Binding Profiling Libr
Yeasen/翌圣生物 品牌
生产厂家厂商性质
上海市所在地
Hieff NGS® In-Situ DNA Binding Profiling Library Prep Kit for Illumina® V2 是针对Illumina®高通量测序平台研发的用于CUT&Tag实验的文库构建试剂盒,适用于100-100,000个细胞起始量的样本建库。CUT&Tag是研究蛋白与DNA互作的技术,相较于传统的ChIP-seq、CUT&RUN,该技术具有文库构建时长更短(仅需7小时)、操作更简单、对起始样本要求更低、抗体投入量更少、文库产量更高等优点。经过细胞捕获、一抗孵育、二抗孵育、转座酶孵育、转座酶激活、掺入DNA标准品、细胞裂解、磁珠回收gDNA、文库扩增和磁珠分选等步骤,靶蛋白结合的DNA片段最终转化为适用于Illumina®平台测序的文库。
本试剂盒包含两个独立模块:BOX-I和BOX-II。BOX-I包含结合细胞的ConA Beads、提取DNA 的DNA Extract Beads以及文库纯化的DNA Selection Beads,BOX-II包含细胞透化剂、抗体结合buffer、转座酶结合buffer、蛋白酶K以及后续文库扩增所需的所有试剂。此外,本试剂盒已在不同种类细胞(如293T、K562、CHO、ESC细胞等)中进行了验证,均具有良好的建库效率和建库产量。本试剂盒提供的所有试剂都经过严格的质量控制和功能验证,大程度上保证了文库的稳定性和重复性。
产品名称 | 产品编号 | 规格 |
Hieff NGS® In-Situ DNA Binding Profiling Library Prep Kit for Illumina® V2 CUT&Tag试剂盒(pA/G-Tn5版) | 12597ES04 | 4 T |
12597ES12 | 12 T | |
12597ES48 | 48 T |
组分编号 | 组分名称 | 12597ES04 | 12597ES12 | 12597ES48 | ||
BOX-I | 12597-A |
| ConA Beads | 40 μL | 120 μL | 480 μL |
12597-B |
| DNA Extract Beads | 280 μL | 840 μL | 3.36 mL | |
12597-C |
| DNA Selection Beads | 400 μL | 1.2 mL | 4.8 mL | |
BOX-II | 12597-D |
| 10 × Binding Buffer | 120 μL | 360 μL | 1.44 mL |
12597-E |
| 10 × Wash Buffer | 600 μL | 1.8 mL | 7.2 mL | |
12597-F |
| 5% Digitonin | 45 μL | 135 μL | 540 μL | |
12597-G |
| 50 × PB Buffer | 6 μL | 18 μL | 72 μL | |
12597-H |
| 10 × Dig-300 Buffer | 400 μL | 1.2 mL | 4.8 mL | |
12597-I |
| pA/G-Transposome Mix | 8 μL | 24 μL | 96 μL | |
12597-J |
| 100 × Activating Buffer | 4 μL | 12 μL | 48 μL | |
12597-K |
| 15 × Terminate Solution | 20 μL | 60 μL | 240 μL | |
12597-L |
| 30 × Proteinase K | 10 μL | 30 μL | 120 μL | |
12597-M |
| DNA Spike-in Mix (5 ng/μL) | 4 μL | 12 μL | 48 μL | |
12597-N |
| 2×HiFi Amplification Mix | 100 μL | 300 μL | 1.2 mL | |
12597-O |
| Goat Anti-Rabbit lgG(H+L)* | 2 μL | 6 μL | 24 μL | |
12597-P |
| N5 (N501)** | 4 μL | -- | -- | |
12597-Q |
| N7 (N701)** | 4 μL | -- | -- | |
注意:*本试剂盒中包含的二抗为山羊抗兔二抗,适用于使用一抗来源于兔源的研究,若需要一抗来源于鼠源的研究,需客户自行准备抗鼠二抗(Yeasen cat#34851ES)或其他等效产品。
**测序时 Index 序列 填写时,测序平台为NovaSeq 6000 v1.0 reagents, MiSeq, HiSeq 2000/2500,则N501-CTCTCTAT,N701-TAAGGCGA。测序平台为NovaSeq 6000 v1.5 reagents,MiniSeq, NextSeq,HiSeq 3000/4000,则N501-ATAGAGAG,N701-TAAGGCGA。
***本试剂盒在实验前需自备蛋白酶抑制剂Cocktail(Yeasen Cat#20123ES)或其他等效产品。
BOX-I:2-8℃保存,BOX-II:-25~-15℃保存,有效期1年。
关于操作
1. 为了您的安全和健康,请穿实验服并戴一次性手套操作。
2. 请于使用前将试剂盒各组分置于室温解冻。解冻后上下颠倒数次充分混匀,短暂离心后置于冰上待用。
3. 推荐在带热盖的PCR仪中进行各步骤反应,使用前应预热PCR仪至反应温度附近。
4. PCR产物因操作不当极容易产生气溶胶污染,进而影响实验结果准确性。推荐将PCR反应体系配制区和PCR产物纯化检测区进行强制性的物理隔离、配备文库构建专用移液器等设备以及定时对各实验区域进行清洁(推荐使用ThermoFisher公司的DNAZapTM高效核酸去除喷雾),以保证实验环境的洁净度。
5. 操作细胞应尽量轻柔以保持细胞活性。
6. 本产品仅用作科研用途!
应用范围
本试剂盒适用于细胞投入量为100-100,000个的CUT&Tag研究。原则上,所有真核生物的新鲜样本或冻存样本都能适用于本试剂盒,但不同的样本类型需要进行不同的样本前处理(如组织样本的单细胞悬液制备、植物样本或者真菌样本的原生质体制备或细胞核悬液制备)后才能按照本实验操作流程进行实验。
ConA beads操作注意事项
1. 使用前将磁珠平衡至室温,请勿将磁珠置于0℃以下存放。
2. ConA beads与细胞结合后,应避免剧烈震荡或用力吹打磁珠-细胞复合物使细胞应力损伤导致磁珠与细胞分离。
3. 在处理磁珠溶液时应尽量避免高转速离心或长时间置于磁力架上,人为造成磁珠凝集。
4. 避免磁珠长时间暴露在空气中使得磁珠干裂或者磁珠-细胞复合物损伤(不超过3 min)。
5. 孵育过程中出现部分磁珠沾壁/聚集是正常现象,只有保证磁珠-细胞复合物处于溶液浸润的范围内,不会影响后续的实验结果。
6. 本产品仅用作科研用途!
关于DNA Extract Beads及DNA Selection Beads操作注意事项
1. DNA Extract Beads用于转座反应后的基因组DNA 提取,DNA Selection Beads在文库纯化过程中使用,切勿弄错。
2. 磁珠使用前应先平衡至室温,否则会导致得率下降、分选效果不佳。
3. 请勿将磁珠置于0℃以下存放。
4. 磁珠每次使用前都应充分振荡混匀或使用移液器上下吹打充分混匀。
5. 转移上清时,请勿吸取到磁珠,即使枪头吸取微量磁珠都将影响后续文库质量。
6. 磁珠洗涤使用的80%乙醇应现用现配,否则将影响回收效率。
7. 产物洗脱前应将磁珠置于室温干燥。干燥不充分容易造成无水乙醇残留影响后续反应;过分干燥又会导致磁珠开裂进而降低纯化得率。通常情况下,室温干燥3 min足以让磁珠充分干燥。
8. DNA纯化或长度分选产物如需保存,可使用0.1×TE Buffer洗脱,产物于4°C可保存2天,-20°C可保存1个月。
关于试剂
1. 不同的Buffer试剂应注意保存条件,避免失效。
2. Digitonin有细胞毒性,且容易降解。在溶液配制过程中请做好个人防护。加入Digitonin的溶液应现配现用,在4℃放置不超过2天。
文库结构
Index 2 (i5)
5’-AATGATACGGCGACCACCGAGATCTACACIIIIIIIITCGTCGGCAGCGTCAGATGTGTATAAGAGACAG-NNNNNN-CTGTCTCTTATACACATCTCCGAGCCCACGAGACIIIIIIIIATCTCGTATGCCGTCTTCTGCTTG-3’
Index 1 (i7)
IIIIIIII: Index 2 (i5), 8 bases;IIIIIIII: Index 1 (i7), 8 bases;-NNNNNN-: 插入序列。
关于抗体选择
CUT&Tag实验中使用的一抗,建议使用ChIP级别或者CUT&Tag、CUT&RUN实验验证过的抗体,若无法达到抗体要求级别,可以使用IF级别抗体进行实验尝试。
实验中建议设置阳性对照和阴性对照组,阳性对照推荐使用样本中表达丰度较高的组蛋白,阴性对照推荐使用不加入一抗但正常加入二抗和转座子,用于判断整个实验过程中是否存在异常,加入非特异性IgG的阴性对照不是必须的,在测序分析中并未提供有价值的信息,可以根据实验需要,自行选择是否添加。
CUT&Tag实验中使用的二抗,建议根据一抗种属以及Protein A/G的亲和能力选择无任何标记或修饰(HRP、FITC等)的二抗,二抗种属来源以及与Protein A/G的亲和能力进行选择,可以参照以下表格:
【注】:+++++代表亲和能力非常强,+++代表亲和能力中等,+代表亲合能力很弱,-代表没有亲合能力。
 二抗推荐:Guinea Pig anti-Rabbit IgG (Heavy & Light Chain) antibody(antibodies-online Cat#ABIN101961)
二抗推荐:Guinea Pig anti-Rabbit IgG (Heavy & Light Chain) antibody(antibodies-online Cat#ABIN101961)
 驴抗兔IgG H&L (Abcam #ab6701)
驴抗兔IgG H&L (Abcam #ab6701)
山羊抗兔IgG H&L(Abcam #ab6702)
山羊抗兔IgG H&L(Yeasen #34850ES)

 Rabbit anti-Mouse IgG (Heavy & Light Chain) Antibody(antibodies-online Cat#ABIN101785)
Rabbit anti-Mouse IgG (Heavy & Light Chain) Antibody(antibodies-online Cat#ABIN101785)
山羊抗小鼠IgG H&L(Abcam #ab6708)
山羊抗小鼠IgG H&L(Yeasen #34851ES)
关于文库扩增
1. 本试剂盒中的文库扩增组分由本公司高保真DNA聚合酶所组成,大大增强了扩增的均一性,即使是低拷贝的基因,也能进行无偏好性地扩增。
2. 推荐使用本公司的Hieff NGS® Tagment Index Kit for Illumina® (Yeasen Cat#12416)模块进行文库扩增,该模块提供了384种不同组合的双端index文库制备引物,大程度上保证了文库构建的稳定性和重复性。
3. 对于PCR循环数,原则上在满足上机的前提下,尽量降低扩增循环数。特别是少量细胞和低丰度蛋白,循环数不足,将导致文库产量低;循环数过多,将导致文库偏好性增加、重复度增加、大片段增多、嵌合产物增加、扩增突变累积等多种不良后果。表2以293T细胞为例,使用中高丰度表达的组蛋白(如H3K4me3、 H3K27me3)进行CUT&Tag实验,细胞投入量、扩增循环数与文库产量之间的关系如下表所示:
表2细胞投入量与扩增循环数推荐表(H3K4me3/H3K27me3)*
细胞投入量 | PCR循环数 | 文库产量 |
100,000 | 11-13 | 10-50 ng/μL |
10,000 | 12-14 | |
1,000 | 15-17 | |
100 | 18-20 |
【注】:*由于不同靶蛋白的表达量、DNA结合能力差异较大。实验中需根据建库起始细胞量、蛋白类型、细胞类型和样本处理情况适当调整扩增循环数。推荐在PCR之前使用qPCR的方法对转座酶插入的片段进行初步定量判断再决定循环数。
本试剂盒适用的细胞投入量为100-100,000个,受细胞类型、抗体选择以及目的蛋白表达丰度的影响,实验中可以兼容的细胞投入量并不是固定的,早期实验时建议投入10,000-100,000个细胞,待获得较理想的结果后,再探索低起始细胞量,细胞量低于10 K时,由于细胞容易损失,实验成功率会降低,建议增加重复组数量。
关于DNA标准品
DNA Spike-in Mix(5 ng/μL)是来源于大肠杆菌 Lambda DNA的三段序列,长度分别为230 bp、250 bp和300 bp,摩尔浓度比约为1:3:10(见图1)。标准品主要用于在不同处理条件下或者不同细胞状态下测序数据Normalization和定量分析。本试剂盒中提供的DNA Spike-in浓度为5 ng/μL,推荐添加量为5 pg/10万(每10 w 细胞建议用TE buffer稀释1000倍加1 μL),根据实际细胞投入量将DNA Spike-in梯度稀释后添加,也可根据靶蛋白的结合DNA能力和丰度自行调整。标准品序列:
Spike-in-1: GTTCCACTCCTGAAGTGTCAAGTACATCGCAAAGTCTCCGCAA
TTACACGCAAGAAAAAACCGCCATCAGGCGGCTTGGTGTTCTTTCAGTTCTT
CAATTCGAATATTGGTTACGTCTGCATGTGCTATCTGCGCCCATATCATCCAGT
GGTCGTAGCAGTCGTTGATGTTCTCCGCTTCGATAACTCTGTTGAATGGCTCT 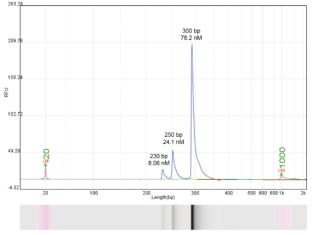
CCATTCCATTCTCCTGTGACTCGGAAGT
Spike-in-2: AGTCGGTGTGAATCCCATCAGCGTTACCGTTTCGCGGTGCTTC
TTCAGTACGCTACGGCAAATGTCATCGACGTTTTTATCCGGAAACTGCTGTCT ![]()
GGCTTTTTTTGATTTCAGAATTAGCCTGACGGGCAATGCTGCGAAGGGCGTTT
TCCTGCTGAGGTGTCATTGAACAAGTCCCATGTCGGCAAGCATAAGCACACA
GAATATGAAGCCCGCTGCCAGAAAAATGCATTCCGTGGTTGTCATACCT
Spike-in-3: CCTTACTGGAATCGATGGTGTCTCCGGTGTGAAAGAACACCAACAGGGGTGTTACCACTACCGCAGGAAAAGGAGGACGTGTGGCGAGACAGCGACGAAGTATCACCGACATAATCTGCGAAAACTGCAAATACCTTCCAACGAAACGCACCAGAAATAAACCCAAGCCAATCCCAAAAGAATCTGACGTAAAAACCTTCAACTACACGGCTCACCTGTGGGATATCCGGTGGCTAAGACGTCGTGCGAGGAAAACAAGGTGATTGACCAAAATCGAAGTTACGAACAAGAAAGCGTCGA