其他品牌 品牌
代理商厂商性质
北京市所在地
高保真基因文库制备仪

基因组测序中的“暗区”和未知区域问题
由于各种原因,所有基因组都包含“难以测序”的区域:
·测序深度不足
·绘图质量差
·分阶段
·病毒、转基因和 CRISPR 编辑的未知整合位点
长基因测序的必要性
基因组编辑在研究、医疗保健和农业方面有着强大的应用。然而,基因组编辑可能导致的各种分子反应事件的范围被低估了,而且该技术在目标位点内外仍然无法预测。这对于为治疗性基因编辑、农业和其他应用提供安全方法具有相当大的影响。预测和验证基因组编辑的结果对于所有应用的成功至关重要。
Cas9 诱导的双链断裂导致一系列预期和意外的结果。 zui 终的编辑结果导致小的 insert、删除或染色体易位或不完整的模板整合。
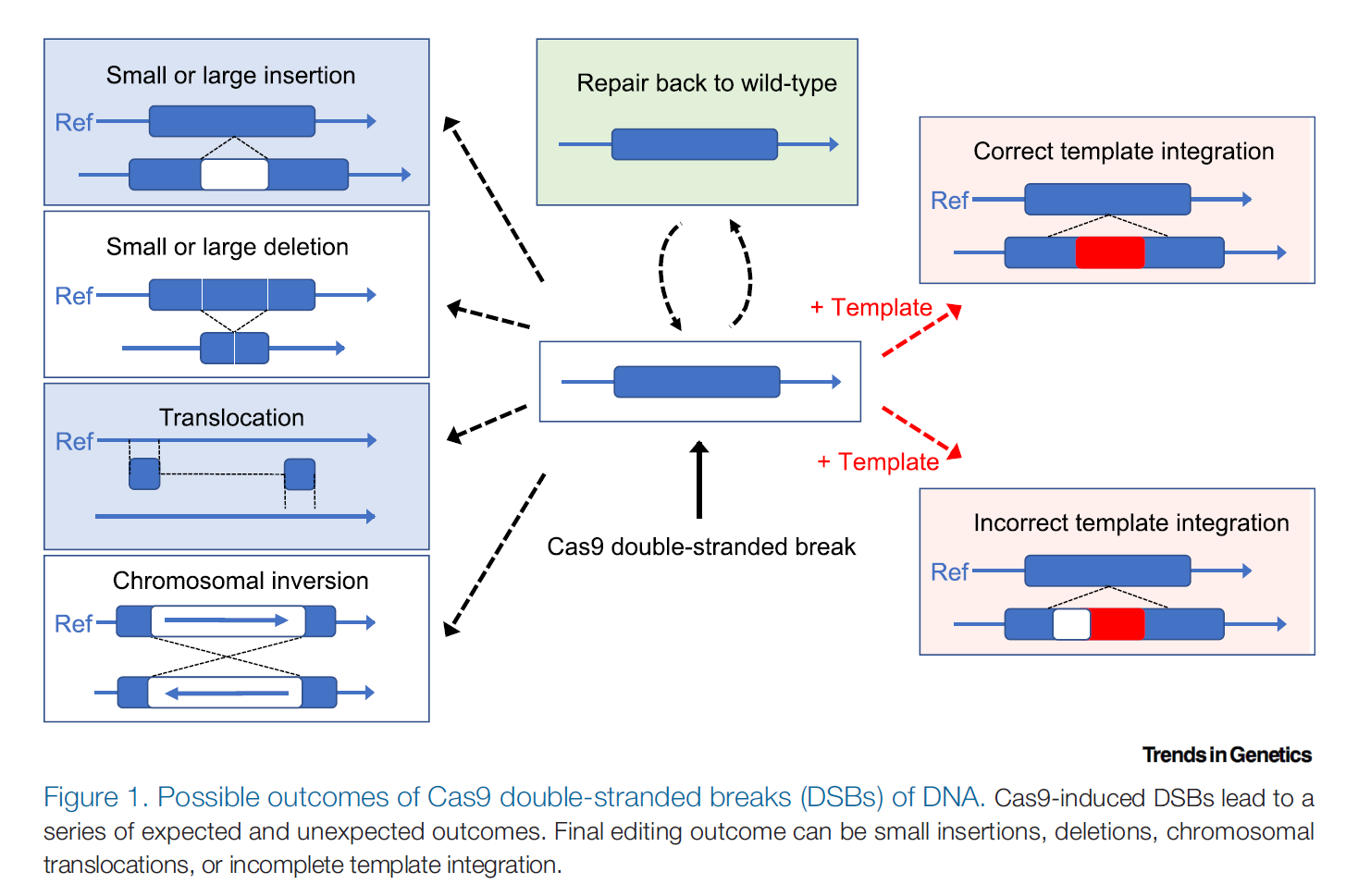
通过各种努力,我们已经采用了多种方法来验证正确的基因编辑并确保未发生针对目标的意外编辑事件。 zui 常用的分子方法是通过 Sanger直接DNA 测序或 PCR扩增子的 NGS ,其大小通常小于 1000 bp。
其他常见方法包括 T7核酸内切酶1 (T7E1) 错配检测分析、通过分解跟踪 insert 缺失 (TIDE) 分析和通过扩增子分析 (IDAA) 检测 insert 缺失、clone PCR 扩增子然后测序,但偶尔也包括全基因组测序(WGS)、阵列比较基因组杂交 (CGH)、Southern 印迹、Fiber-FISH 和 FISH。
大多数情况下,这些方法仅提供有关基因编辑位点周围有限区域的信息,不会捕获来自 CRISPR/Cas诱变效应的整个序列,尤其是不会检测更大的缺失。为此Kosicki 等人在Nat. Biotechnol上发表的《Repair of double-strand breaks induced by CRISPR–Cas9 leads to large deletions and complex rearrangements》论文,强调了将分子分析扩展到包括 CRISPR-Cas9 诱导的 DSB 位点周围的几千碱基的重要性,并展示了长读长测序如何可以高分辨率的分析基因编辑的准确性。
长读长测序可以解决许多这些挑战,但在常规实验室分析中大规模应用不切实际且成本高昂。
Xdrop高保真长基因文库制备仪
Samplix 的 Xdrop™ 仪器可以捕获基因组 DNA 的长 (>100 kb) 区域。 使用 Xdrop 为科学家提供了目标富集的经证实的好处(减少时间、成本和数据分析),与已建立的目标富集方法相比具有额外的优势:
解锁基因组的任何部分
·以仅 100-200 bp 的序列信息为目标的任何区域
快速适应新目标
·识别和捕获新目标,在 4-5 天内准备好进行测序
从您的长读仪器中获得更多收益
·测序能力集中在您感兴趣的区域
减少测序伪影
·使用无 PCR、无偏差的目标扩增方法
Samplix Xdrop™用于富集长基因组DNA 片段,以通过长读长和短读长测序分析 CRISPR-Cas9 编辑细胞中的基因型和等位基因状态 。在论文《Verification of CRISPR editing and finding transgenic inserts by Xdrop Indirect sequence capture followed by short- and long- read sequencing》中,研究人员通过Xdrop技术在一组 CRISPR 修饰的诱导多能干 (iPS) 细胞系中检测到 CRISPR-Cas9 诱导的意外基因编辑。 尽管使用其他几种互补方法进行了全面分析,但早期验证 CRISPR 编辑的尝试并未检测到这些意外编辑。
在这项研究中,他们还证明用于评估 CRISPR-Cas9 基因组编辑事件的基于 PCR 的标准程序无法提供足够准确的验证。CRISPR 编辑的验证应基于对更大区域的检查。
Xdrop方法所检测序列的位置可能与主要感兴趣区域(如基因编辑位点)相距 5-10 千碱基 (kb) 或更远。
其实验过程是在将高分子量 (HMW) DNA 、PCR 试剂和引物分配到双乳液液滴内并进行反应后,含有目标 DNA 的液滴通过液滴 PCR识别,然后用 DNA 嵌入荧光染料进行染色。
只有含有来自感兴趣区域 (ROI) 的 DNA 的液滴才会产生检测序列扩增子并发出荧光。由于双乳液液滴的大小和组成,这些可以在标准流式细胞仪细胞的分选仪器上进行分类,以富集荧光液滴。
然后分离含有长 DNA 片段的液滴,并通过液滴中的单分子多重置换扩增(dMDA) 进行扩增。dMDA 的扩增能力能够从 6 pg 的输入 DNA 中产生约 1.5 μg 的扩增 DNA,并且对大于 5 kb 的分子 zui 有效。由此产生的富集 DNA 与短读长和长读长测序平台兼容。
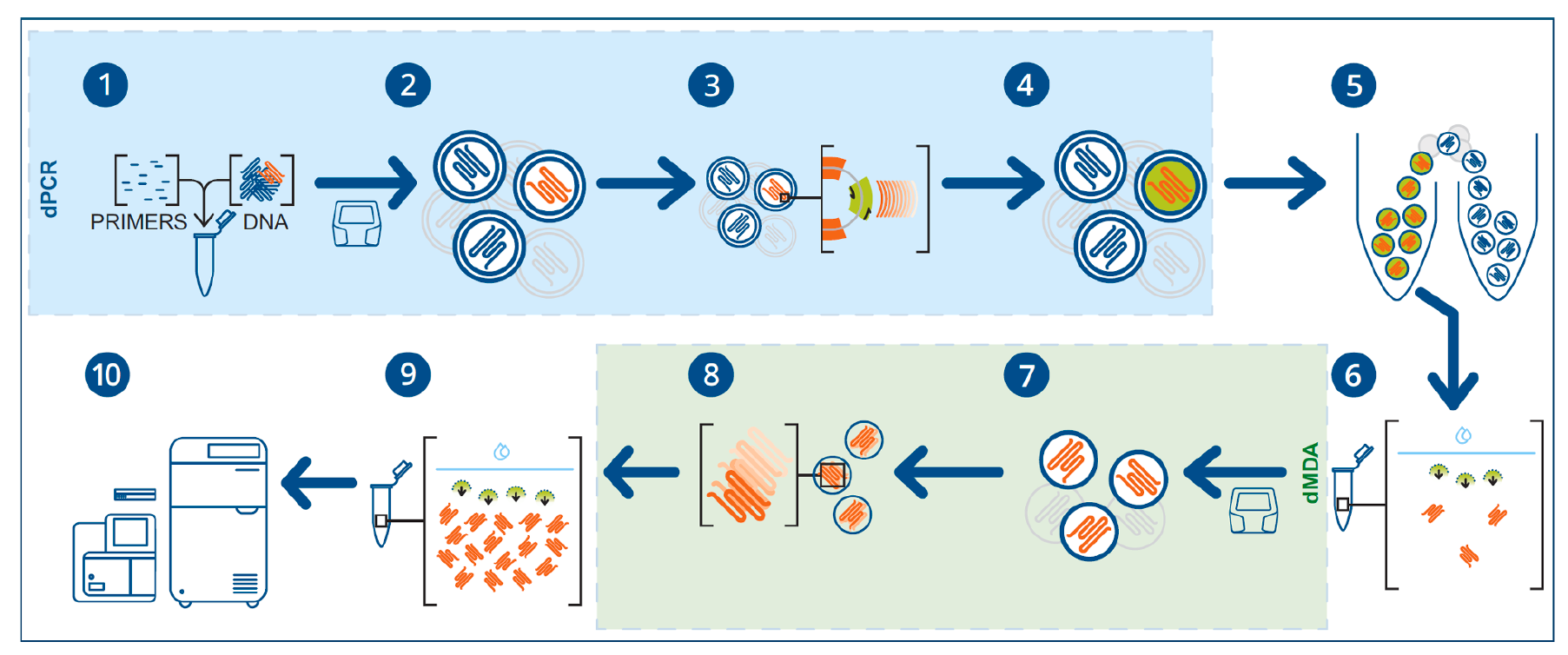
Xdrop间接序列捕获方法可采用距基因编辑位点 5–10 kb 或更远的距离设计的引物,它在高深度测序提供了大约 100 kb 长的区域的序列读取覆盖。这允许对基因编辑站点周围区域中潜在的意外编辑进行完整调查。Xdrop技术展示了如何在事先不了解其基因组整合的情况下也能够识别转基因 insert 位点。
细胞治疗,细胞疗法,细胞杀伤,免疫疗法,杀伤细胞,细胞免疫,哺乳动物细胞,微生物细胞,单细胞,细胞功能,基因测序,细胞基因组学,单细胞测序、Sanger测序、NGS、PCR、 T7核酸内切酶1错配检测分析、跟踪 insert 缺失TIDE、扩增子分析 (IDAA) 检测 insert 缺失、全基因组测序WGS、比较基因组杂交 CGH、Southern 印迹、Fiber-FISH、FISH、CRISPR/Cas